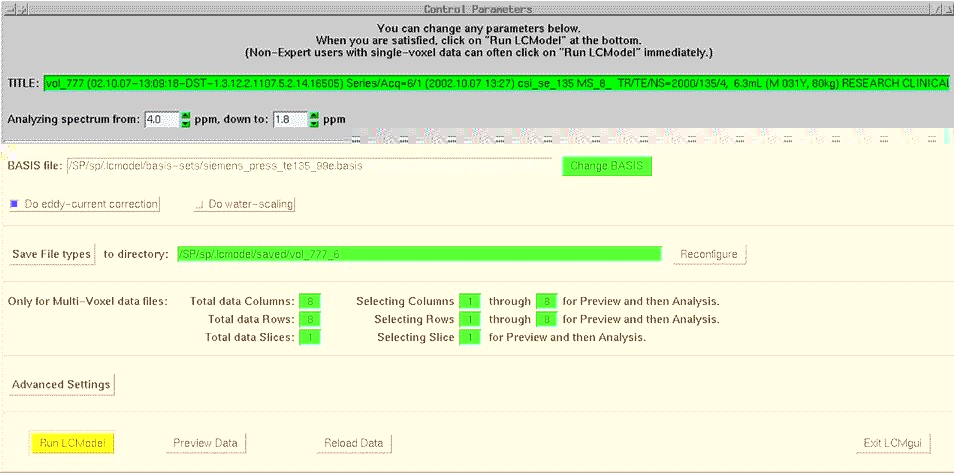
対応システム

Linux(Red Hat系Linux推奨。CentOS4,5,6,7で動作検証)
Intel PentiumII以上のCPUを搭載したPCが必要になります。
Window ManagerはKDE, GNOME, fvwm2を推奨します。
Old GNOME Window Managerをご使用になるとLCMgui windowを手動でMoving, Resizingする必要があります。
PentiumIV 2GHz以上のPCをご使用になれば、今まで使用していたUnixよりも解析が早くなると思います(開発者より)。
※ PostScriptファイルを表示するビューアーをインストールしてください。また、ネットワークから データ取得する場合、Samba, NFS,
FTP等をインストールすることを推奨します(環境によって異なります)。
弊社では、LCModelをプリインストールしたLinuxシステムも販売しておりますので、お気軽にお問い合わせください。
データ入出力対応ファイルフォーマット
入力対応ファイルフォーマット(PressまたはSteamシークエンスのRAWデータ)Single-voxel data
- Bruker fid files (if necessary, converted by you to analog mode with Bruker’s convdta). Also for ParaVision-3 fid files
- GE 5.x Probe raw P-files and spectrum G-files
- GE Probe raw P-files, including (new) 14.0 M3 P-files. Also for P-files with multi-channel (phased-array) data
- Philips SDAT/SPAR pairs and enhanced DICOM files.
- Picker (later Marconi, now Philips) DUMP files
- Siemens *rda files transferred from the syngo PC console and Siemens raw files from the (old) Numaris-3 Unix console
- Toshiba(CANON) rawData files and (new) Version 7.xx (and later) DICOM files
- Varian/Agilent fid files
一部のMulti-voxel(CSI) data(Philips, Siemensでは検証済み GEについてはSage側で対応必要)
※上記ファイルフォーマットに関しては、各MRIメーカーにご確認ください。
※MRI側に保存されているMRSデータの保存先は各MRIメーカーにご確認ください。
※日立製MRに関しては、メーカー側ソフトウェアで対応しており弊社のLCModelでは解析できません。
出力対応ファイルファーマット(標準)
- PostScript(スペクトル形状を含む画像ファイル)
- CSV(Excel形式のテキストの代謝物濃度の解析結果)
- TXT(プレーンテキストの代謝物濃度の解析結果)
※処理スクリプトの設定によりLinux変換ツールを自動起動してPDF、BMPなどにも変換できます。
※DICOMファイル対応にはオプションのPACSソフトウェアをご利用ください。
Basis-setsについて
脳MRS解析時に成分の基準となるBasis-setファイルが必要になります。
現状、対応している条件は下表になります。Pressの場合、記載されていない条件でもシミュレーションにより開発元で作成できます。Steamの場合はご相談ください。
※ Version 6.3 よりMRI装置メーカーごとのBasis-setは無くなり各メーカー共通のBasis-setになりました。
| 磁場強度 | シークエンス | エコー時間(TE) |
|---|---|---|
| 1.5T | Press | 30, 35, 60, 135, 144, 270, 288 |
| 3.0T | Press | 25, 30, 35, 60, 80, 135, 144, 270, 288 |
- 備考1:
- 脳MRS解析(脳内のコリン、クレアチン、Nアセチルアスパラギン酸などの解析)では、Basis-setファイル(MRS解析の基準となるファントム撮像から作成された化合物のスペクトルデータのセット)が必要となります。
- 備考2:
- TRについては10000msを推奨していますが、一般には3000ms~4000ms程度が使用されています。
- 備考3:
- 肝臓脂肪、筋細胞脂肪、乳腺脂肪の解析の場合は内部シミュレーションで行いますので必要ありません(ソフトの仕様上、ダミーファイルを選択する必要はありますが解析に使用しません)。
ネットワーク接続例
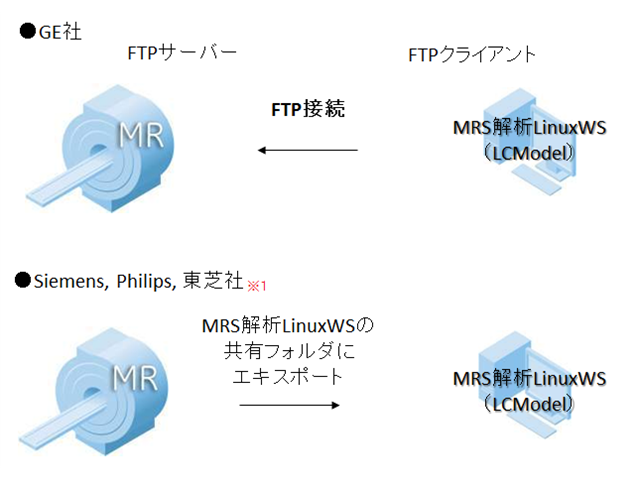
※ネットワークを介してのデータ取得は、基本MRIコンソールとの接続のみの対応となります。
※ネットワーク接続に関しては、ネットワーク管理者、各MRIメーカーにご確認ください。
※ 分析結果ファイルをネットワークプリンタ等にプリントアウトする場合、プリンタメーカーからLinux用ドライバーが提供されている必要があります。
※1キヤノン(東芝)装置とネットワーク接続するためにはDICOM通信可能なソフトウェアを介在させる必要があります。
※LCModel解析結果をDICOM化して、PACSなどに送信するには付属のオプションソフトウェアをご利用ください。
解析対象代謝物一覧
脳内代謝物 (デフォルトおよびCSFモード)
- Ala
- L-Alanine
- Asp
- Asparate
- Cho
- Choline
- Cr,Cre
- Creatine
- GABA
- γ-Aminobutyric Acid
- Glc
- Glucose
- Gln
- Glutamine
- Glu
- Glutamate
- GPC
- Glycerophosphocholine
- GSH(Ver6.3で追加)
- Glutathione
- PCh
- Phosphocholine
- PCr
- Phosphocreatine
- Ins,mI
- myo-Inositol
- Lac
- L-Lactate
- NAA
- N-AcetylAsparate
- NAAG
- N-AcetylAspartylGlutamate
- Scyllo
- scyllo-Inositol
- Tau
- Taurine
- -CrCH2
- Creatine methylene group
- Gua(Ver6.3で削除)
- Guanidinoacetate
2HG(2-hydroxyglutarate)解析に関してはVer6.3以降で使用できる専用のbasis-setがあります。
※ LCModelによる2HG解析の参照論文(2HG)
筋細胞脂肪(Ver 6.1追加 muscleモード)
- IMCL13 (intramyocellular lipids)
- 筋細胞内脂肪の1.3ppm付近のメチレン(-CH2-)ピーク
- EMCL15 (extramyocellular lipids)
- 筋細胞外脂肪の1.5ppm付近のメチレン(-CH2-)ピーク
脂肪 (lipid,liver,breast モード)
- Lip13
- 脂肪酸内のメチレンプロトン -CH2- に対応した1.3ppm付近のピーク
- Lip09
- 脂肪酸の末端メチルプロトン CH3-に対応した0.9ppm付近のピーク
- Lip16
- –CH2–O–CO–CH2–CH2– 脂肪酸内のカルボキシル基のβ位のメチレンプロトン
- Lip21
- –CH2–CH2–CH=CH– 不飽和脂肪酸内のビニレン基のα位のメチレンプロトン
- Lip23
- CH2–O–CO–CH2–CH2– 脂肪酸内のカルボキシル基のα位のメチレンプロトン
- Lip28
- –CH=CH–CH2–CH=CH– 不飽和脂肪酸内の2つのビニレン基の間のα位のメチレンプロトン
- Lip41
- –CH2–O–CO–CH2–CH2– グリセリン内のメチレンプロトン
- Lip43
- –CH2–O–CO–CH2–CH2– グリセリン内のメチレンプロトン
- Lip52,
Lip53 - –CH=CH– 不飽和脂肪酸内のビニレンプロトン
および >CH–CH2–O–CO–CH2–CH2– グリセリン内のメチンプロトン - Cho
- コリンに対応した3.2ppm付近のピーク
- Glycg
- (liver-11のみ)グリコーゲンに対応した3.5–3.9ppm付近のピーク