LCModel
-1H MRスペクトルから代謝産物の濃度を自動計算-
※ 2021/02 に、開発者のDr. Stephen Provencherより健康上の理由で引退と、LCModel開発中止及びHP上でのオープンソース化の連絡がありました。
エルエイシステムズでの製品版LCModelソフトウェアのライセンス販売は終了しますが、日本国内でのLCModelセットアップ済みPC販売、および有償での保守、サポートは継続しております。
今後、bin2rawなどのバージョンアップ、他OSでの動作版の開発、カスタムbasis-setの作成などのサポート業務も行っています。
初期導入支援サービス
お客様に代わって導入時のセットアップ作業、サポートを実施致します
また、その他MRSデータの処理に関するご相談もお受け致します
お気軽にお問合せ下さい
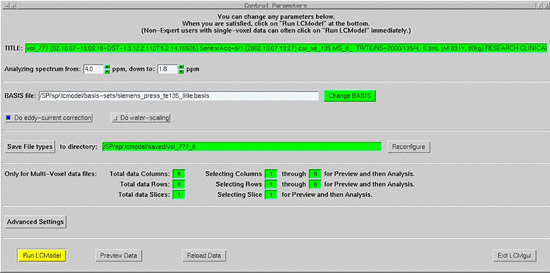
導入セットアップ作業:
LCModelの導入には、LCModelソフトウェアをインストールする以外にも以下の作業が必要になります
- Linux環境の構築、Linuxターミナルでのコマンドラインインストール
- 使用するMR装置の測定条件(磁場強度、PressTE)に対応したbasis-setファイル(代謝物毎の標準スペクトル内部データベース) のインストール
測定対象(筋脂肪、肝臓脂肪、2HG、MegaPress等)により以下の設定が必要になる場合があります
- コントロールパラメータの設定、プロファイルの作成、専用basis-setの入手
また、MRS解析結果(スペクトル、解析数値)の処理のために以下のようなファイルを出力するカスタム設定も可能になります
- 解析結果の画像ファイル化、PDF化(標準ではPostScriptファイルのみ)
- 測定結果のテキスト化、スペクトルグラフ座標データの取得
- バッチファイルによる多数データの連続処理
| サービス名 | 導入セットアップ作業 | PC | お問合せ対応 |
|---|---|---|---|
| 標準導入 | △ マニュアルをご用意しますので お客様ご自身でセットアップ頂きます |
× お客様ご自身で ご用意下さい |
メール,電話,リモート対応 |
| セットアップPC 販売 |
〇 | 〇 当社でPCを用意 セットアップします |
メール,電話,リモート対応 現地訪問での初期操作講習 |
| 訪問セットアップ | 〇 | × お客様所有のPCに セットアップします |
メール,電話,リモート対応 現地訪問での初期操作講習 |
年間保守サービス
年間保守サービスをご契約頂くことで、動作環境・操作説明・MR装置アップデート時のbin2raw対応など、LCModelの操作に関するお客様の課題解決を支援させて頂きます
| サービス名 | お問合せ対応 (メール,電話,リモート) |
現地訪問による課題対応 |
|---|---|---|
| 標準保守 | 〇 | × |
| 訪問保守 | 〇 | 〇 但し、1回/年までで、施設内スタッフ交代時の操作講習も可能です |
Basis-setの新規作成等、特別な技術検討が必要となる場合は別途お見積りさせて頂く場合があります
追加での現地訪問が必要になる場合は別途お見積りにより対応させて頂きます
LCModelとは
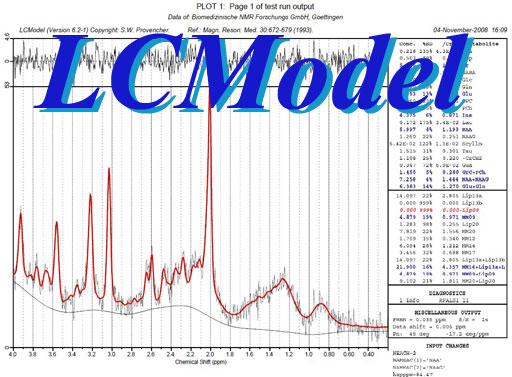
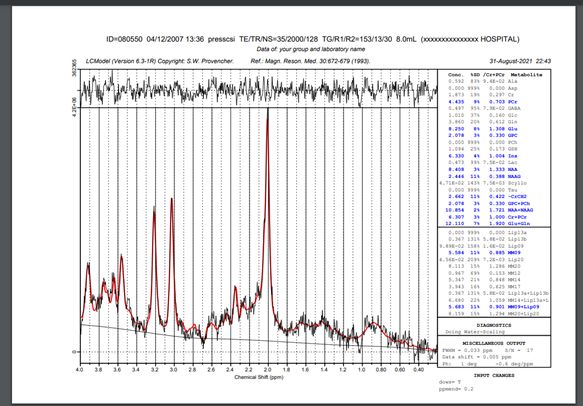
LCModel はMRI装置の1H MRS(PressまたはSteamシークエンス)から代謝産物の定量解析を行うソフトウェアです。代表的なMRメーカーのrawデータの読み込みに対応 しており、スペクトルを自動処理(FT, 位相補正等)し、カーブフィットした後に、MR装置、TE毎のBasis-setデータと比較し、代謝物毎のピーク分離で計算されるピークエリアから各代 謝産物の定量(水分を基準としたmM(mmol/L)濃度、または対Creatine比)計算を自動的に行います。分析結果はMRスペクトル、代謝産物の 名称、濃度、標準偏差(SD)の情報と共にポストスクリプト(PS)ファイル、CSVファイル、TXTファイルへ出力されます。オプション設定で指定代謝 物のピーク分離解析結果を出力することもできます。また解析後プロセス用のシェルスクリプトを変更してPSファイルからの画像変換、集計などをカスタマイ ズできます。
・Ver6.1からIMCL (筋細胞内脂肪)、EMCL (筋細胞外脂肪)の解析機能が追加されました(適用例:スポーツ医学、生活習慣病などの研究・解析)。
・Ver6.2から、脂肪(lipid)、肝脂肪(liver)、胸部脂肪(breast)(適用例:メタボリック症候群、肝脂肪などの研究・解析)と脳髄 液(CSF:通常のNAA,Cho,Crのピークの代わりに、CSFではLac,Glcのピークを基準に解析します) 解析が追加されました。 脂肪解析の場合はbasis-setは必要ありません。(2007年9月)
※ 2021/2/10に最終バージョンとなるVer 6.3-1Rがリリースされました。
※ 2020/5/30にVer 6.3-1Pがリリースされました。GE社、Bruker社の新ファイル形式読込に対応しています。Toshiba表記がCanon/Toshibaに変更されています。解析プログラムは6.3-1Lと変更ありません。
※ 2019/3/15にVer 6.3-1Nがリリースされました。GE社DV27のPファイルの読み込みに対応しています。解析プログラムは6.3-1Lと変更ありません。
※ 本ソフトウェアを医療及び研究用途にご使用になる場合、ユーザの責任の下でご利用ください。
※ 記載内容は予告なしに変更する場合があります。
※ 以下 LCModel home page上にダミーライセンス設定方法、製品版でのcontrol parameterでのライセンス認証の無効化方法が記載されており、標準的な医療用MR用の脳basis-setファイルへのリンクがあります。
解析対象代謝物一覧
対応システム
データ入出力対応ファイルフォーマット
Basis-setsについて
ネットワーク接続例
ダウンロード、インストール
PDFカタログ
FAQ – よくある質問についての回答
☆オプション ソフトウェア☆
1.LCModel with ![]() プラットフォーム(DICOM送受信、データベース、ビューア、レポート)
プラットフォーム(DICOM送受信、データベース、ビューア、レポート)
<重要> GE社のMRでLCModelをご利用の皆様へ
GE社のソフトウェアをDV26以降にバージョンアップする場合、先にLCModelのバージョンを6.3-1L(最終版6.3-1R)以上にアップグレードしてください。6.3-1Hより古いバージョンではDV26以降に対応していないのでLCModelで解析ができません。
LCModel最新バージョンのダウンロードはこちらから行ってください。
GE社でのDV27以降のソフト、Airコイルの組み合わせでの測定で、正常なMRSのPファイルが出力されない現象が報告されています。LCModel側では対処できない原因のため、GE社のサービスの方と測定時の設定について相談してください。
解析対象代謝物一覧
脳内代謝物 (デフォルトおよびCSFモード)
| Ala | L-Alanine |
| Asp | Asparate |
| Cho | Choline |
| Cr,Cre | Creatine |
| GABA | γ-Aminobutyric Acid |
| Glc | Glucose |
| Gln | Glutamine |
| Glu | Glutamate |
| GPC | Glycerophosphocholine |
| GSH(Ver6.3で追加) | Glutathione |
| PCh | Phosphocholine |
| PCr | Phosphocreatine |
| Ins,mI | myo-Inositol |
| Lac | L-Lactate |
| NAA | N-AcetylAsparate |
| NAAG | N-AcetylAspartylGlutamate |
| Scyllo | scyllo-Inositol |
| Tau | Taurine |
| -CrCH2 | Creatine methylene group |
| Gua(Ver6.3で削除) | Guanidinoacetate |
2HG(2-hydroxyglutarate)解析に関してはVer6.3以降で使用できる専用のbasis-setがあります。
※ LCModelによる2HG解析の参照論文(2HG) クリック
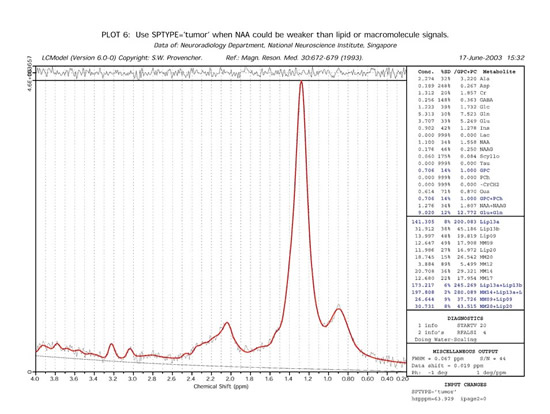
筋細胞脂肪(Ver 6.1追加 muscleモード)
| IMCL13 (intramyocellular lipids) | 筋細胞内脂肪の1.3ppm付近のメチレン(-CH2-)ピーク |
| EMCL15 (extramyocellular lipids) | 筋細胞外脂肪の1.5ppm付近のメチレン(-CH2-)ピーク |
脂肪 (lipid,liver,breast モード)
| Lip13 | 脂肪酸内のメチレンプロトン -CH2- に対応した1.3ppm付近のピーク |
| Lip09 | 脂肪酸の末端メチルプロトン CH3-に対応した0.9ppm付近のピーク |
| Lip16 | –CH2–O–CO–CH2–CH2– 脂肪酸内のカルボキシル基のβ位のメチレンプロトン |
| Lip21 | –CH2–CH2–CH=CH– 不飽和脂肪酸内のビニレン基のα位のメチレンプロトン |
| Lip23 | CH2–O–CO–CH2–CH2– 脂肪酸内のカルボキシル基のα位のメチレンプロトン |
| Lip28 | –CH=CH–CH2–CH=CH– 不飽和脂肪酸内の2つのビニレン基の間のα位のメチレンプロトン |
| Lip41 | –CH2–O–CO–CH2–CH2– グリセリン内のメチレンプロトン |
| Lip43 | –CH2–O–CO–CH2–CH2– グリセリン内のメチレンプロトン |
| Lip52, Lip53 |
–CH=CH– 不飽和脂肪酸内のビニレンプロトン および >CH–CH2–O–CO–CH2–CH2– グリセリン内のメチンプロトン |
| Cho | コリンに対応した3.2ppm付近のピーク | Glycg | (liver-11のみ)グリコーゲンに対応した3.5–3.9ppm付近のピーク |
対応システム

Linux(Red Hat系Linux推奨。CentOS4,5,6,7で動作検証)
Intel PentiumII以上のCPUを搭載したPCが必要になります。
Window ManagerはKDE, GNOME, fvwm2を推奨します。
Old GNOME Window Managerをご使用になるとLCMgui windowを手動でMoving, Resizingする必要があります。
PentiumIV 2GHz以上のPCをご使用になれば、今まで使用していたUnixよりも解析が早くなると思います(開発者より)。
※ PostScriptファイルを表示するビューアーをインストールしてください。また、ネットワークから データ取得する場合、Samba, NFS, FTP等をインストールすることを推奨します(環境によって異なります)。
弊社では、LCModelをプリインストールしたLinuxシステムも販売しておりますので、お気軽にお問い合わせください。
データ入出力対応ファイルフォーマット
●入力対応ファイルフォーマット(PressまたはSteamシークエンスのRAWデータ)
Single-voxel data
- Bruker fid files (if necessary, converted by you to analog mode with Bruker’s convdta). Also for ParaVision-3 fid files
- GE 5.x Probe raw P-files and spectrum G-files
- GE Probe raw P-files, including (new) 14.0 M3 P-files. Also for P-files with multi-channel (phased-array) data
- Philips SDAT/SPAR pairs and enhanced DICOM files.
- Picker (later Marconi, now Philips) DUMP files
- Siemens *rda files transferred from the syngo PC console and Siemens raw files from the (old) Numaris-3 Unix console
- Toshiba(CANON) rawData files and (new) Version 7.xx (and later) DICOM files
- Varian/Agilent
fidfiles
一部のMulti-voxel(CSI) data(Philips, Siemensでは検証済み GEについてはSage側で対応必要)
※上記ファイルフォーマットに関しては、各MRIメーカーにご確認ください。
※MRI側に保存されているMRSデータの保存先は、各MRIメーカーにご確認ください。
※日立製MRに関しては、メーカー側ソフトウェアで対応しており弊社のLCModelでは解析できません。
●出力対応ファイルファーマット(標準)
- PostScript(スペクトル形状を含む画像ファイル)
- CSV(Excel形式のテキストの代謝物濃度の解析結果)
- TXT(プレーンテキストの代謝物濃度の解析結果)
※処理スクリプトの設定によりLinux変換ツールを自動起動してPDF、BMPなどにも変換できます。
※DICOMファイル対応にはオプションのPACSソフトウェアをご利用ください。
Basis-setsについて
・脳MRS解析時に成分の基準となるBasis-setファイルが必要になります。
・現状、対応している条件は下表になります。Pressの場合、記載されていない条件でもシミュレーションにより開発元で作成できます。Steamの場合はご相談ください。
※ Version 6.3 よりMRI装置メーカーごとのBasis-setは無くなり各メーカー共通のBasis-setになりました。
| 磁場強度 | シークエンス | エコー時間(TE) |
| 1.5T | Press | 30, 35, 60, 135, 144, 270, 288 |
| 3.0T | Press | 25, 30, 35, 60, 80, 135, 144, 270, 288 |
備考1: 脳MRS解析(脳内のコリン、クレアチン、Nアセチルアスパラギン酸などの解析)では、Basis-setファイル(MRS解析の基準となるファントム撮像から作成された化合物のスペクトルデータのセット)が必要となります。
備考2: TRについては10000msを推奨していますが、一般には3000ms~4000ms程度が使用されています。
備考3: 肝臓脂肪、筋細胞脂肪、乳腺脂肪の解析の場合は内部シミュレーションで行いますので必要ありません(ソフトの仕様上、ダミーファイルを選択する必要はありますが解析に使用しません)。
ネットワーク接続例
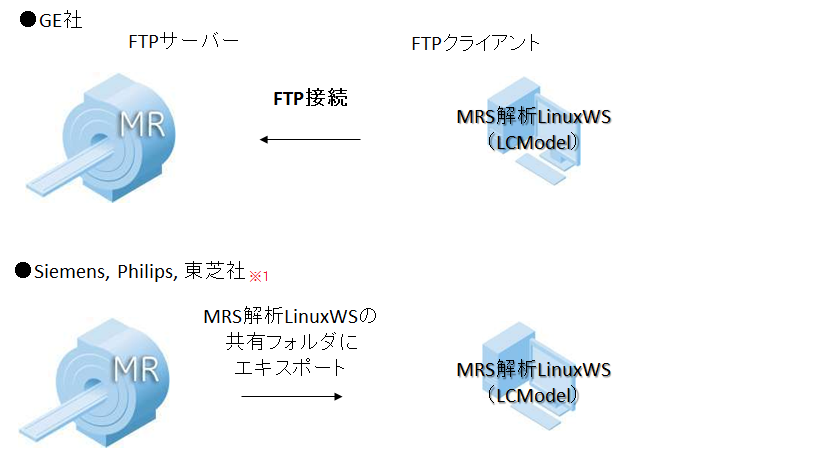
※ ネットワークを介してのデータ取得は、基本MRIコンソールとの接続のみの対応となります。
※ ネットワーク接続に関しては、ネットワーク管理者、各MRIメーカーにご確認ください。
※ 分析結果ファイルをネットワークプリンタ等にプリントアウトする場合、プリンタメーカーからLinux用ドライバーが提供されている必要があります。
※1キヤノン(東芝)装置とネットワーク接続するためにはDICOM通信可能なソフトウェアを介在させる必要があります。
※LCModel解析結果をDICOM化して、PACSなどに送信するには付属のオプションソフトウェアをご利用ください。
ダウンロード、インストール
(既存のユーザは上書きインストールで最新版にアップデートできます)
ダウンロード:
LastUpdate(2021/02/07 LastVersion) :
64bit版 lcm-64_v6.3-1R-20210207.tar (11.4Mbyte)
32bit版 lcm-32_v6.3-1R-20210207.tar (7.6Mbyte)
※64bit版はGE社DV16のみ読み込み不具合があります。DV16を読み込む場合は32bit版を使用するか、64bit版インストール後に32bit版tarファイルを解凍して内部から~/gelx/bin2rawファイルをコピーして使用して下さい
※アップデート後GEデータを解析する時、新しいデータのタグ情報と既存のProfileに保存されているタグ情報との互換性がない場合、保存ディレクトリがsaved直下になる場合があります。(解析時にタグ情報から自動で作成される保存ディレクトリが作成されない)
その場合GUI画面から「Reconfigure」ボタンを選択して「Restore Default」でProfileのディレクトリ生成方法をリセットし、
解析終了時に「Exit&Save」を選択してProfileを保存してください
オプション ソフトウェア
LCModel with  プラットフォーム
プラットフォーム
仮想化テクノロジーを利用して1台のPCでLinux OS上でLCModel解析、そして、Windows OS上では高機能プラットフォームソフトウェアMyrian® Advance(DICOM通信/データベース管理、マルチモダリティビューア、レポート作成)を実行します。
<操作例>
 | ||||
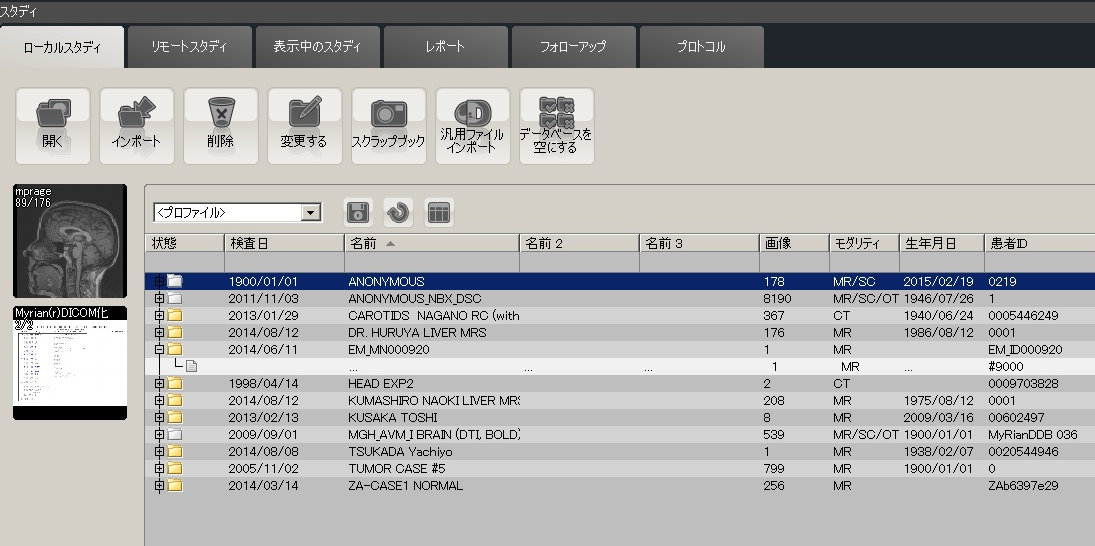
| ① Dicom MRSデータ受信 (MR装置により未対応) |
 |
|||
 | ||||
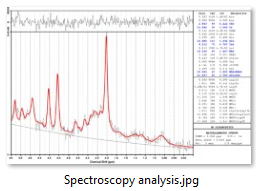
| ②Export→LCModel解析 |  |
|||
 | ||||
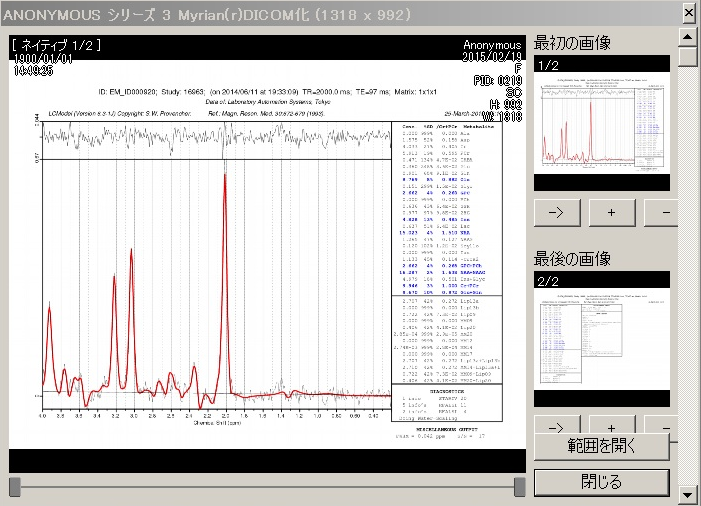
| ③ Dicom化(ヘッダー編集可) |  |
|||
 |
④’印刷/保存/PACS送信 | |||
 |
 | |||
| ④ 編集と保存 |  |
|||
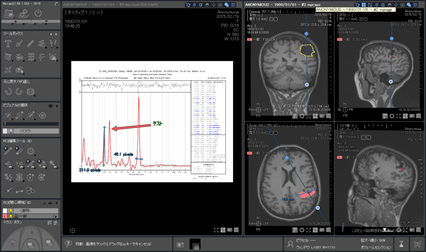 | ||||
| ⑤ レポート作成 |  |
|||
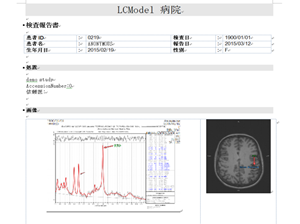 | ||||
| ⑥ レポート印刷/保存/PACS送信 |  |
|||
 | ||||
- LCModelフリーウェア版(ライセンス認証無効設定版)を納入PCにプリインストールします。
- すでにLCModel搭載PCをご購入のお客様にはPCのセンドバックで対応致します。ご相談ください。

Webアプリ(Linuxサーバ)版 LCModel
-Linux版LCModel のコマンドラインでの実行をブラウザGUIで操作-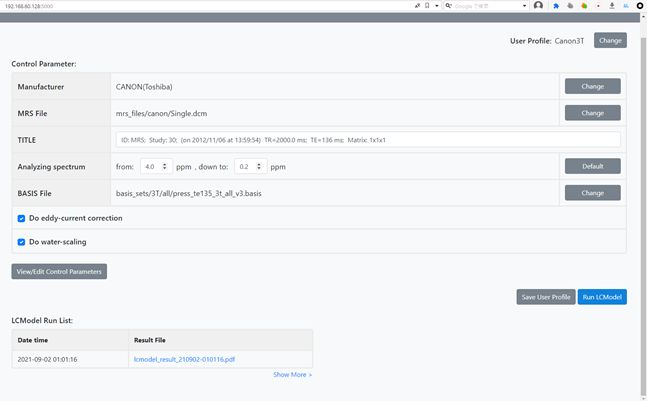
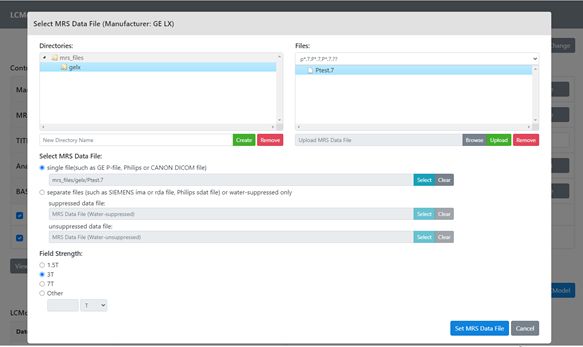
LinuxにインストールしたLCModel本体を、Python Webサーバ(Ubuntu20.04)からコマンドラインで動作させ、 Webブラウザから操作可能なWebインターフェースのアプリを開発しています。
Webアプリ(Linuxサーバ)版 LCModelカタログこのページのトップへ